UNCOVERING THE IN SILICO REGULATORY POTENTIAL OF MICROSATELLITE SEQUENCES IN GLYCINE MAX (L.) MERR.
UNCOVERING THE IN SILICO REGULATORY POTENTIAL OF MICROSATELLITE SEQUENCES IN GLYCINE MAX (L.) MERR.
Abstract
The aim of our work was to identify the regulatory potential of microsatellite noncoding DNA in relation to gene expression. SSRs (Simple Sequence Repeats) are currently used for gene mapping and search, parentage determination, and barcoding systems. For a long time, it was believed that these sequences are not involved in the inheritance of a trait or its regulation. However, to date, some mechanisms for the influence of non-coding sequences on the regulation of gene expression have been identified. One such mechanism is DNA regions that are capable of binding to transcription factors called enhancers. Non-coding regions of the plant genome, particularly soybean, are understudied, and the total number of discovered enhancer sequences is very small. Bioinformatic analysis of known SSRs in Glycine max allows in silico detection of putative regulatory regions of the genome. The present information indicates the relevance of the topic. In this research, using the iEnhancer-2L algorithm, 101 possible enhancers were found among the 935 SSRs in the SOYBASE database, from which 8 SSRs with numerous strong enhancers were selected. Among these SSRs, Unipro UGENE and the Sitecon package identified 6 that had consensus sites with transcription binding site factors. The data obtained will be useful not only theoretically, but also as a strategy to accelerate the selection process. Due to the possibility of finding mechanisms to influence the sites that regulate the expression level of genes responsible for useful traits.
1. Введение
Сателлитная ДНК состоит из множества тандемных повторов, которые играют важную роль в клеточных процессах, включая сегрегацию хромосом, организацию генома и защиту концевых участков хромосом. Большинство повторов сателлитной ДНК имеют либо нуклеосомную длину, либо 5–10 п.н. и занимают центромерные, перицентромерные или теломерные области. Для сателлитных ДНК некоторых организмов были идентифицированы несколько консервативных сателлит-специфичных последовательностей, кривизна ДНК, симметрии диад и перевернутые повторы. Последовательности сателлитной ДНК либо встроены в высококомпактный гетерохроматин с низким содержанием генов, либо в специализированный хроматин, отличный от эухроматина. Тем не менее некоторые сателлитные ДНК транскрибируются в некодирующие РНК, которые могут играть важную роль в генной регуляции. Сателлитная ДНК является одной из наиболее эволюционно-изменчивых структур, что обуславливает высокую видовую специфичность , .
Сателлитная ДНК классифицируется на микросателлиты: это короткие повторяющиеся последовательности ДНК, длина которых варьируется от 2 до 5 нуклеотидов, минисателлиты – это более длинные повторяющиеся последовательности ДНК, длина которых может варьироваться от 10 до 100 нуклеотидов и макросателлиты – это самый длинный тип сателлитной ДНК с повторяющимися единицами длиной от 100 до нескольких тысяч нуклеотидов , . Характерным представителем микросателлитной ДНК является SSR.
SSR (Simple Sequence Repeats) или по другому называемые у человека STR (Short Tandem Repeats) — это повторяющиеся короткие не кодирующие последовательности генома. Свойство микросателлитов накапливать большое количество нуклеотидных замен обуславливает разный вид и длину схожих участков у разных представителей одного вида, что позволяет использовать сателлитную ДНК в криминалистике, для определения родства, составления генеалогического древа, а также при картировании генов
, . Долгое время функции не кодирующей части ДНК были неизвестны из-за чего эти участки назывались «мусорными», но по мере развития геномики обнаруживались новые функции и шло разделение её на разные виды: теломеры, мобильные элементы, интроны, инсуляторы, участки, регулирующие экспрессию генов, некоторые из них имеют вирусное происхождение в виде остаточных последовательностей ДНК .SSR вызывают большой интерес у исследователей, поскольку могут быть использованы для выявления генетических вариаций между разными растениями, а также для изучения эволюции различных видов. SSR-локусы полезны для понимания генетики признаков растений, таких как устойчивость к болезням, засухоустойчивость и адаптация к изменениям окружающей среды. Они также могут помочь понять механизмы реагирования растений на различные условия окружающей среды и как они взаимодействуют с другими организмами в своих экосистемах , , .
Известно, что некоторые мутации микросателлитов способны стать причиной заболеваний. Имеется информация об оказании влияния на процесс сплайсинга
, и на экспрессию генов . Среди функций не кодирующей части ДНК, следует особо отметить регуляторную, поскольку изменение уровня экспрессии определенных генов позволяет улучшать хозяйственно-ценные признаки. Участки, обладающие данной функцией, называются энхансерами и сайленсерами, усиливающими и подавляющими экспрессию генов соответственно , .Энхансеры — это особые участки генома, действующие как сайты связывания специфичных для гена факторов транскрипции. В отличие от промоторов, энхансеры могут находиться на большом расстоянии от гена или даже находиться на другой хромосоме от него. По степени влияния на транскрипцию гена энхансеры могут быть разделены на несколько типов, сильные, сбалансированные, слабые и не активные .
Микросателлитная ДНК может являться энхансером у растений. Ранее исследователи уже использовали SSR для регуляции экспрессии определенных генов в растениях. Например, анализировали микросателлиты участвующие в повышении устойчивости Arabidopsis thaliana за счет повышения их сопротивления к водному стрессу .
SSR имеют различные мотивы, знания о которых может быть использовано для создания искусственных энхансеров в растениях путем введения этих последовательностей ДНК в геном растения. Это позволит увеличивать эксперессию гена-мишени у растения путем вставки специфичных для энхансеров мотивов рядом с геном .
Знания о последовательностях генома, отвечающих за регуляцию экспрессии определённых генов могут помочь в селекционной работе, позволяя отбирать для дальнейшей гибридизации наиболее подходящие генотипы. Поэтому в нынешний период развития пост-геномных технологий, где требуется решение современных задач в сельском хозяйстве, знание и понимание функционального назначения того или иного генетического конструкта могут оказать решающую роль.
В данном исследовании была поставлена цель — спрогнозировать энхансерный потенциал наиболее изученных и популярных SSR-локусов и определить их сайты связывания с транскрипционными факторами.
2. Методы и принципы исследования
Для проведения анализа из открытой базы данных о геноме сои министерства сельского хозяйства США "SOYBASE.ORG" была взята информация о нуклеотидных последовательностях известных микросателлитных локусах. Критерием отбора локусов являлось наличие информации о первичной последовательности ДНК. Для идентификации последовательности как энхансер, а также для определения силы этой последовательности был использован сервис iEnhancer-2L.
iEnhancer-2L — инструмент на основе PseKNC (Pseudo K-tuple Nucleotide Composition/Псевдо К-картежный нуклеотидный состав) выполняющий двухуровневое предсказание, на первом этапе которого производятся расчёты того является ли последовательность поступившая на входе энхансером, после чего, в случае положительного результата, программа переходит на второй этап, а именно на проверку силы предсказанного энхансера. Работает данная программа на основе машинного обучения по эталонным данным методом «складного ножа». Обучающий набор данных, включал информацию о 742 сильных энхансеров, 370517 слабых энхансеров и 5257994 неэнхансеров. На основание этого обучающегося набора, данный сервис строит прогноз о возможном наличии энхансера в качестве подстроки в предоставленной последовательности. Данные, поступившие на вход программы в формате FASTA или в виде нуклеотидной последовательности разбиваются на отрезки по 200 н.п. и с шагом в 1 н.п. идет их анализ. Последовательности длинной менее 200 н.п. распознаются как не энхансер .
Для определения возможных сайтов связывания транскрипционных факторов на обнаруженных энхансерах был использован программный пакет SITECON от Unipro UGENE, основанный на наборе данных о консервативных конформационных и физико-химических свойствах, выявленных на основе анализа наборов известных сайтов связывания. Для анализа была выбрана модель "GATA" представляющая собой семейство факторов транскрипции со способностью связаться с последовательностями GATA в ДНК .
3. Основные результаты
Из базы данных SOYBASE.ORG были отобраны нуклеотидные последовательности 935 микросателлитных локусов, обладающих мотивами AT и ATT. После чего на iEnhancer-2L была проанализирована каждая последовательность. В результате из общего количества микросателлитов были отобраны 101, обладающие возможной энхансерной функцией.
Микросателлиты, среди которых были найдены возможные энхансерные последовательности, были разделены по характерным для них мотивам АТТ и АТ. Для мотива АТТ среди 602 микросателлитных маркеров было найдено 62, содержащих последовательности, имеющие разную теоретическую энхансерную силу (табл. 1).
Таблица 1 - Микросателлитные маркеры с мотивом АТТ (n/m)*
Satt702(18/0) | Satt701(56/77) | Satt687(0/146) | Satt684(0/3) | Satt669(1/0) |
Satt644(0/19) | Satt643(6/58) | Satt597(0/25) | Satt589(0/4) | Satt582(83/35) |
Satt567(0/25) | Satt541(0/2) | Satt538(0/78) | Satt530(0/80) | Satt521(0/19) |
Satt516(0/11) | Satt480(0/70) | Satt475(2/73) | Satt459(39/0) | Satt451(2/40) |
Satt444(0/38) | Satt423(14/22) | Satt400(0/119) | Satt398(0/12) | Satt393(0/14) |
Satt380(0/13) | Satt357(0/153) | Satt356(0/96) | Satt349(0/1) | Satt348(2/0) |
Satt343(0/103) | Satt336(1/0) | Satt329(36/53) | Satt324(34/6) | Satt322(0/94) |
Satt314(9/6) | Satt311(86/22) | Satt305(43/27) | Satt303(0/56) | Satt289(0/29) |
Satt256(0/83) | Satt255(0/216) | Satt240(20/0) | Satt192(0/19) | Satt187(20/42) |
Satt185(0/14) | Satt173(21/65) | Satt165(0/56) | Satt145(28/79) | Satt136(106/35) |
Satt133(12/85) | Satt122(60/80) | Satt119(0/6) | Satt115(51/0) | Satt080(67/58) |
Satt071(18/16) | Satt070(17/124) | Satt052(0/27) | Satt049(16/110) | Satt041(0/88) |
Satt020(0/2) | Satt014(0/6) | - | - | - |
Примечание: * n- количество сильных энхансеров, m-количество слабых энхансеров
Общее количество микросателлитных маркеров с мотивом АТ составило 333, а всего среди них локусов обладающих возможными энхансерными последовательностями 39 (табл. 2).
Таблица 2 - Микросателлитные маркеры с мотивом АТ (n/m)*
Sat_423(0/19) | Sat_420(0/53) | Sat_406(0/5) | Sat_404(7/52) | Sat_392(0/39) |
Sat_391(28/26) | Sat_385(0/72) | Sat_381(28/86) | Sat_366(0/51) | Sat_363(0/9) |
Sat_357(0/66) | Sat_348(28/5) | Sat_341(0/27) | Sat_340(0/62) | Sat_313(25/0) |
Sat_304(0/64) | Sat_298(0/3) | Sat_289(15/15) | Sat_285(0/11) | Sat_272(0/1) |
Sat_265(0/4) | Sat_253(0/88) | Sat_228(0/63) | Sat_217(0/16) | Sat_214(6/85) |
Sat_207(0/16) | Sat_205(14/79) | Sat_182(0/88) | Sat_141(1/0) | Sat_116(26/5) |
Sat_114(20/96) | Sat_100(99/51) | Sat_097(0/27) | Sat_094(10/11) | Sat_084(0/3) |
Sat_81(0/26) | Sat_069(11/11) | Sat_043(0/26) | Sat_036(0/56) | - |
Примечание: n- количество сильных энхансеров, m-количество слабых энхансеров
Среди общего количества микросателлитных маркеров сильными энхансерными последовательностями обладали 42, из которых 28 с мотивом АТТ и 13 с мотивом АТ. Помимо этого, особо выделялись Satt701, Satt582, Satt311, Satt136, Satt122, Satt115, Satt080, sat_100, обладающие большим количеством энхансерных последовательностей (более 50).
SSR обладающие большим количеством сильных энхансерных последовательностей были проанализированы на наличие сайтов связывания транскрипционного фактора нацеленного на мотив GATA с использованием SITECON via Unipro UGENE (табл. 3).
Таблица 3 - Расположение сайтов связывания к транскрипционному фактору в пределах сателлитов
SSR | Регионы | Вероятность связывания % | Сила энхансера |
Sat_100 | 352-377 | 90,17 | Strong/сильный |
349-374 | 80,93 | Strong/сильный | |
217-242 | 80,48 | Weak / Strong Слабый/сильный | |
49-74 | 90,02 | Weak/слабый | |
Satt701 | 448-473 | 79,82 | Not an enhancer |
282-307 | 78,61 | Weak / Strong Слабый/сильный | |
74-99 | 80,78 | Strong/сильный | |
61-86 | 82,46 | Strong/сильный | |
5-30 | 78,16 | Strong/сильный | |
Sat582 | 279-304 | 88,52 | Strong/сильный |
275-300 | 80,24 | Strong/сильный | |
271-296 | 80,75 | Strong/сильный | |
149-174 | 77,46 | Strong/сильный | |
Satt122 | 431-456 | 87,51 | Strong/сильный |
345-370 | 78,76 | Strong/сильный | |
338-363 | 83,05 | Strong/сильный | |
321-346 | 78,81 | Strong/сильный | |
278-303 | 82,73 | Strong/сильный | |
170-195 | 87,16 | Weak/слабый | |
159-184 | 81,13 | Weak/слабый | |
Satt115 | 421-446 | 91,08 | Not an enhancer/ не энхансер |
303-328 | 83,25 | Not an enhancer/ не энхансер | |
290-315 | 80,99 | Not an enhancer/ не энхансер | |
195-220 | 78,75 | Weak / Strong Слабый/сильный | |
95-120 | 87,61 | Not an enhancer/ не энхансер | |
83-108 | 77,66 | Not an enhancer/ не энхансер | |
48-73 | 84,82 | Not an enhancer/ не энхансер | |
8-33 | 80,46 | Not an enhancer/ не энхансер | |
3-28 | 79,53 | Not an enhancer/ не энхансер | |
Satt080 | 481-506 | 79,08 | Weak/слабый |
430-455 | 80,72 | Weak/слабый | |
346-371 | 80,38 | Weak/слабый | |
304-329 | 79,77 | Weak/слабый | |
263-288 | 92,11 | Strong/ сильный | |
220-245 | 82,07 | Strong/ сильный | |
198-223 | 80,74 | Not an enhancer / Strong Не энхансер/ сильный | |
81-106 | 81,23 | Not an enhancer/ не энхансер | |
68-93 | 79,24 | Not an enhancer/ не энхансер |
Из этого числа SSR было обнаружено 6: Sat_100, Satt581, Satt582, Satt122, Satt115, Satt080 имеющие сайты связывания транскрипционных факторов, пересекающиеся с регионами, спрогнозированными как потенциальные энхансеры. Satt311 и Satt136 не имели нуклеотидного консенсуса, что может быть объяснено несоответствием GATA Sitecon с прогнозом IEnhancer-2L. Возможно регуляторный потенциал данных SSR-локусов будет раскрыт в последующих работах с применением других Sitecon моделей.
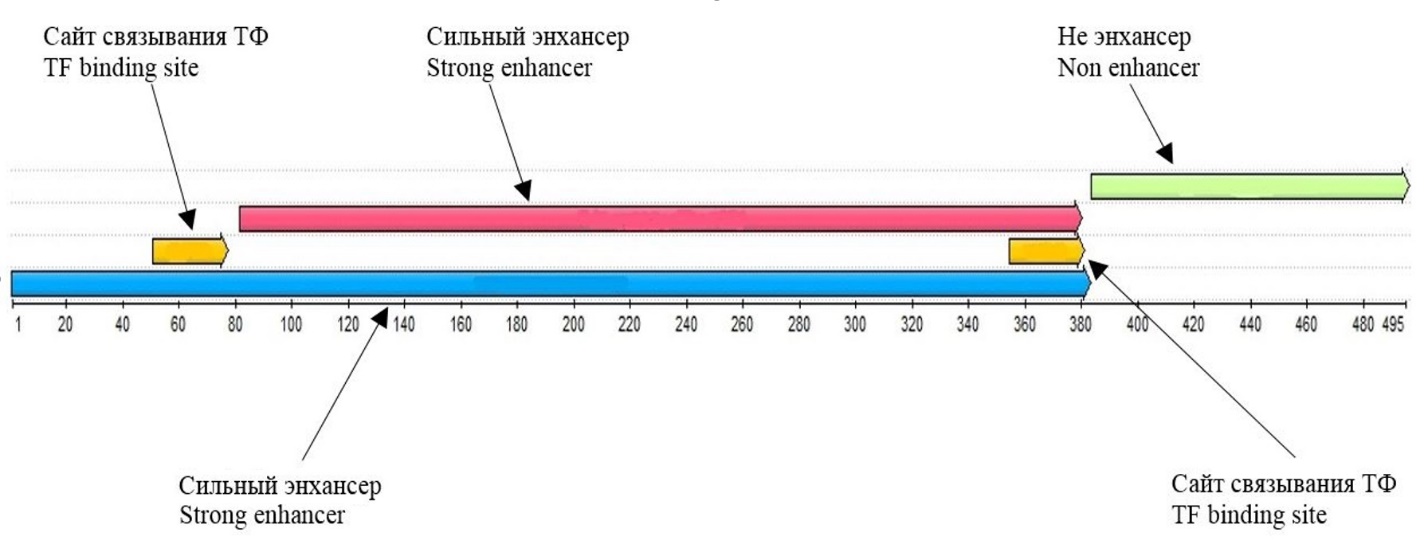
Рисунок 1 - Аннотационная карта микросателлита Sat_100 с длиной 495 п.н
4. Заключение
В числе взятого из базы данных SOUYBASE.ORG общего количества микросателлитных маркеров имеются 42 возможных энхансера, предсказанных iEnhancer-2L. При этом среди микросателлитов с мотивом АТТ предполагается энхансерная активность у 45%, у мотива АТ — 33%. При этом отличающихся по большому количеству именно сильных последовательностей было 7 микросателлитных маркеров, из которых 6 имели мотив АТТ и 1 — мотив АТ. По нашему мнению, имеется взаимосвязь между тандемной повторностью и силой энхансера. Так, последовательности с мотивом АТТ имели большее количество сайтов GATA и, как правило, обладали большей силой и соотношением энхансеров к не-энхансерам. Известные микросателлитные локусы, чья функция до сих пор не описывалась, могут иметь регуляторную функцию: усиливать экспрессию генов. На следующих этапах следует проводить поиск предполагаемых генов-мишеней для этих локусов с целью экспериментально подтвердить регуляторное влияние SSR-локусов на экспрессию генов. Для экспериментального подтверждения проведённых исследований требуется использование методик, таких как иммунопреципитация хроматина с последующим секвенированием, полногеномное картирование модификаций гистонов. Как результат биоинформатическое прогнозирование функций SSR — актуальное направление для исследований, ввиду возможности предварительного скрининга и отсеивания тех последовательностей чья функция выходит за рамки позитивной регуляции экспрессии генов.
