Comparative analysis of the nucleotide and amino acid sequence of the chymosin protein in the mammalian evolutionary series
Comparative analysis of the nucleotide and amino acid sequence of the chymosin protein in the mammalian evolutionary series
Abstract
The article presents the results of comparative analysis of amino acid and nucleotide sequence of chymosin exons in the region of the catalytic centre (position 25-220) of mammals of different habitats with different diets using phylogenetic analysis tools. Data on nucleotide and amino acid sequence alignment of chymosin exons were obtained, conserved and variable regions were determined, pairwise comparative analysis of nucleotide and amino acid identity was performed, and the spectrum of amino acid occurrence was determined for the protein as a whole and in the region of the catalytic centre. In the course of phylogenetic analysis, the dendrogram constructed for the nucleotide sequence of the chymosin gene was used to identify clades of six groups, which were formed by representatives of the families analysed. The data obtained can be used to predict the digestive and immune functions of the protein at the cellular level.
1. Введение
Химозин (Chn, EC 3.4.23.4), неонатальная желудочная аспарагиновая пепсиноподобная протеаза молодых млекопитающих, вызывает свертывание молока посредством расщепления пептидных связей κ-казеина (κCS) на участке Met105-Phe106 и применяется для производства сыров , , , . Фактически, использование Chn при производстве сыра считается одним из самых ранних биологических применений ферментов, а остатки сыра датируются примерно 3000-2800 годами до н.э. , , , . Chn синтезируется как пре-прохимозин, который расщепляется до зрелого белка Chn за счет отщепления 16-ти аминокислот, который образуют гидрофобный сигнальный пептид , .
Клеточные сигнальные аспарагиновые протеазы (APS) повсеместно распространены в природе и выполняют разнообразные функции от пищеварения до участия в формировании иммунитета , , , . APS разделены на различные семейства на основе гомологии, структуры и чувствительности к пепстатину , . APS включают в себя пять надсемейств и 16 семейств. Большинство принадлежат к семейству A1 – пепсиноподобных APS. Структурно пепсиноподобные AP обычно определяются двумя гомологичными доменами, которые охватывают два каталитических остатка аспарагиновой кислоты в триплете Asp-Thr/Sergly , , , .
Хотя роль Chn в свертывании молока и производстве сыра хорошо известна, в то же время анализ эволюционной динамики в ряду позвоночных аминокислотной и нуклеотидной последовательности крайне актуальна, в связи с тем, что Chn был обнаружен у большого числа видов, как наземных, так и водных млекопитающих , . Анализ аминокислотных последовательностей и кодирующих последовательностей белков (экзонов) занимает центральное место в изучении молекулярной эволюции. Среди основных инструментов — построение филогенетических деревьев, которые собираются на основе алгоритмов, учитывающих количество несовпадающих аминокислот или оснований между выровненными последовательностями.
Обоснования исследования белков заключаются в наличии механизмов мутаций и последующего отбора, которые приводят к изменению в кодирующей последовательности гена и, соответственно, в аминокислотной последовательности белка в процессе эволюции , , , . Так, в частности, дупликация и делеция генов являются мощными драйверами эволюционных изменений, которые иллюстрируются изменчивым ферментативным спектром, участвующим в переваривании белков позвоночных, включая Chn, и являются реакцией на общие факторы окружающей среды (среда обитания, терморегуляция, рацион питания) .
Потеря генов долгое время считалась второстепенной движущей силой адаптивной эволюции. Тем не менее, в настоящее время потеря генов рассматривается в качестве ключевого фактора в формировании эволюционных изменений , . Генетический репертуар генов желудка в ряду позвоночных, является хорошим примером ведущей роли потери генов в адаптивной фенотипической изменчивости , , . Анализ последовательности гена и первичной структуры аспарагиновых протеиназ птиц и млекопитающих (куриный эмбриональный пепсиноген, ренин и пепсиноген человека, химозин быка) выявил сходство в организации генов, и эти результаты подтверждают мнение о том, что эти гены ферментов произошли от общего предкового гена. Напротив, в аспарагиновых протеиназах S. cerevisiae, C. tropicalis, M. pusillus и M. Miekhey, в первичной структуре интрон не обнаружен. Однако в генах аспарагиновой протеиназы Rhizopus niveus и аспарагиноврй протеиназы A. awamori были обнаружены 1 и 3 коротких интронах соответственно, но их экзон-интронные соединения находились в других положениях, нежели в генах аспарагиновых протеиназ млекопитающих и птиц . Семейство генов пепсина широко распространено, но распределено хаотично в ряду млекопитающих, подвержено псевдогенизации, и как указывалось выше, потери гена , . Несмотря на псевдогенный статус у человека, Chn обнаружен у нескольких видов млекопитающих, рептилий и птиц, костистых рыб , , у приматов, кожнокрылых, грызунов, китообразных и непарнокопытных . Chn является псевдогеном у человека за счет сдвига рамки считывания и последующего преждевременного стоп-кодона, вызванного делецией в экзоне 4 .
В отличие от других членов семейства пепсинов, мало что известно об эволюционной истории, распространении и функции Chn в линиях млекопитающих, несмотря на то, что Chn демонстрирует консервативную четвертичную структуру и каталитические остатки, он проявляет необычный профиль с низкой общей протеолитической активностью и высокой специфичностью к κ-казеину молока , , , . Так, в частности, филогенетический анализ на основе ДНК и аминокислотных последовательностях гена Chn девятнадцати видов млекопитающих, рыб, птиц, рептилий и грызунов, выявил идентичность в диапазоне от 64% до 98% , . Аминокислотный сравнительный анализ рекомбинантного химозина (rChn) марала, быка и верблюда выявил, что последовательности зрелых химозинов оказались идентичными на 92,6%. rChn быка и верблюда имеют схожие третичные структуры, при этом идентичность их первичных структур составляет всего 84,8%. rChn быка и марала содержит больше отрицательно заряженных аминокислот . Построена модель пространственной структуры rChn лося с позиции топографии положительных и отрицательных поверхностных зарядов в сравнении с rChn коровы и верблюда. Ранее исследователи получили и изучили rChn овцы (Ovis aries), козы (Capra hircus), буйвола (Bubalus arnee bubalis) и верблюда (Camelus dromedarius) , , , . Позднее были получены и изучены rChn яка (Bos grunniens), альпаки (Vicugna pacos) и алтайского марала (Cervus elaphus sibiricus) , , .
Повторяющаяся инактивация Chn раскрывает эволюционные взаимодействия между пищеварительной и иммунной системами, регулируемого псевдогенизацией . Роль Chn в передаче IgG изучалась исключительно у жвачных животных, Chn расщепляет κCS и запускает высвобождение сывороточного белка, который содержит IgG, в кишечный тракт для неселективного всасывания в течение первых часов после рождения , , , .
Картина псевдогенизации гена Chn, по-видимому, совпадает с приобретением новых стратегий передачи иммунитета, как ранее предполагалось для плотоядных животных, приматов и грызунов, за исключением жвачных животных , , , . Хотя активность Chn напрямую связана со свертываемостью молока, она коррелирует с переносом иммуноглобулинов, зависящим от молозива и наличия ингибиторов трипсина в молозиве, что обеспечивает дополнительную защиту от ферментативного переваривания в тонком кишечнике , , . В связи с этим было высказано предположение, что такое видоспецифичное распределение является результатом диетической адаптации, результатом приобретения пассивного переноса материнского иммунного иммуноглобулина G (IgG) – «иммунная гипотеза». Однако стратегии передачи IgG у разных млекопитающих различаются, так в частности у человека, например, IgG передается от матери к плоду на последних стадиях беременности (материнский перенос).
В связи с этим цель нашего исследования – выявить отличительную особенность аминокислотной и нуклеотидной последовательности Chn млекопитающих различных сред обитания, с различным рационом питания используя инструменты филогенетического анализа, для получения данных, которые могут использоваться для прогнозирования функции белка на клеточном уровне.
2. Методы и принципы исследования
Нуклеотидная последовательность экзонов гена Chn была получена из базы данных NCBI в разделе Gene: Camelus bactrianus ferus (XP_006194819.1), Bos Bosgrunniens (JX839990.1), Bos Taurus (NP851337.1), Bubalus bubalis (XP006065015.1), Ovis aries (NP001009804.1), Capra hircus (NP001272688.1), Camelus dromedaries (NP_001290503.1), Rattus norvegicus (NP_064476.1), Cervus canadensis (MT225406.1), Delphinapterus leucas (XM_022583844.1), Tupaia chinensis (XM_006149562.1), Vicugna pacos (XM_031680278.1), Alces alces alces (MT542132.1), Capreolus pygargus (WGN96205.1). Множественное выравнивание (Multiple Sequence Alignment) нуклеотидных последовательностей проводилось с помощью базы данных EMBL-EBI (Великобритания) во вкладке CLUSTAL OMEGA.
Аминокислотная последовательность экзонов была получена из базы данных UniProt . Выравнивание аминокислотной последовательности производилась с использованием базы данных EMBL-EBI: Множественное выравнивание проводили во вкладке Multiple Sequence Alignment в разделе CLUSTAL OMEGA (Великобритания), парное – во вкладке Pairwise Sequence Alignment в разделе EMBOSS Needle.
Множественное выравнивание гомологичных последовательностей консервативных участков экзонов в области каталитического центра (в положении 25-220) было получено с помощью редактора выравнивания биологических последовательностей BioEdit. Редактирование и улучшение визуализации проводилось во вкладке Graphic View . Полученные данные визуализированы с помощью Excel с цветовым указанием семейств и сравниваемыми представителями семейств.
Филогенетический анализ проводили с использованием программы UniPro UGENE. Для построения филогенетического дерева использована модель Phylip Naighbor-Joining (Phylip Naighbor-Goining model), методом максимального правдоподобия на основе модели Tamura-Nei models , , , принимая во внимание различия в скорости замены между нуклеотидами и неравенство частот нуклеотидов, а также методом Neighbor-Joining method (NJ) для создания филогенетических (эволюционных) и фенетических (сходство на основе признаков) деревьев, на основе матрицы расстояний как метод минимальной эволюции .
Моделирование и визуализация трехмерной структуры. 3D модель химозина Camelus dromedaries построена в программе rcsb.org/3d-view/1CMS.
Статистическая обработка данных и визуализация полученных результатов проводилась с использованием пакетов программного обеспечения Prism 8.0.1 (Graphpad, USA) и Microsoft Excel.
3. Основные результаты и обсуждение
Анализируемые группы животных относятся к классу плацентарных млекопитающих водной и наземной среды обитания (рис. 1). Выбор для исследования представителей семейств определяется тем, что ген Chn данных представителей широко используется в качестве источника рекомбинантного химозина (rChn), ген интегрируется в состав экспрессионных челночных плазмид с дальнейшим включением в геном клеток продуцентов (E.сoli, Pichia pastoris, также известная как Komagataella phaffi, Kluyveromyces Lactis, Aspergillus niger и др.) для синтеза белка Chn , , , .
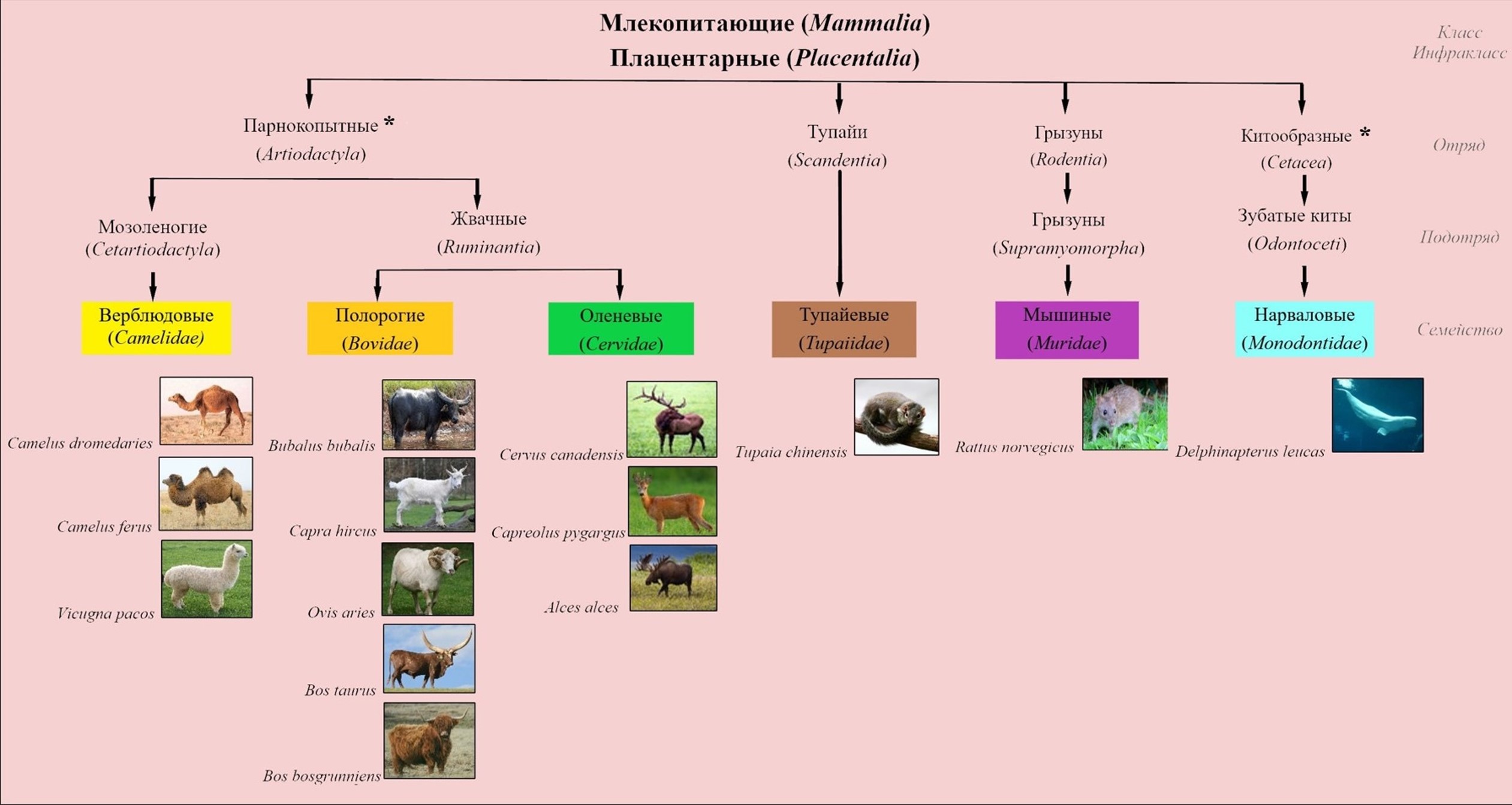
Рисунок 1 - Анализируемые представители семейств класса млекопитающие (Mammalia)
Примечание: * - отряды парнокопытные (Artiodactyla) и китообразные (Cetacea) объединяются в парафилетический отряд китопарнокопытные (Cetartiodactyla)
Наиболее консервативными остатками аминокислот и их позиции, после выравнивания по всем представителям семейств, являются – Lys-Gly-K (26-28), Leu-Arg (30-31), Leu-Lys-Glu (34-36), Gly-Leu-Leu-Glu-Asp-Phe-Leu (38-44), Gln (47), Ser (52), Gly (59), Pro-Leu-Thr (65-67), Tyr-Leu-Asp (69-71), Tyr-Phe-Gly (74-76), Ile-Tyr (78-79), Gly-Thr-Pro (81-83), Gln-Glu-Phe (85-87), Val (89), Phe-Asp-Thr-Gly-Ser-Ser (91-96), Trp-Val-Pro-Ser (99-102), Tyr-Cys (104-105), Ser (107), Val-Cys (109-110), H (113), Arg-Phe-Asp-Pro (115-118), Ser (121), Thr-Phe (123-124), Lys-Pro-Leu (129-131), Tyr-Gly-Thr-Gly-Ser (135-139), Leu (142), Tyr-Asp-Thr-Val-Thr-Val-Ser (146-152), Ile-Val (154-155), Gln-Thr-Val-Gly-Leu-Ser-Thr (159-165), Pro-Gly (168-169), Phe-Thr-Tyr (172-173), Phe-Asp-Gly- Ile-Leu- ly (177-182), Ala-Tyr-Pro (184-186), Ala (188), Tyr-Ser (192-193), Pro (195), Phe-Asp (197-198), Met-Met (200-201), Leu-Val-Ala (205-207), Asp (209), Phe- Ser-Val-Tyr (211-214), Arg (217) и Gln (220). Наибольшее количество замен одной аминокислоты среди анализируемых представителей выявлено у Rattus norvegicus (21 аминокислота, 12%). У Delphinapterus leucas (4 аминокислоты, 2,3%), Tupaia chinensis (3 аминокислоты, 1,7%), Alces alces (1 аминокислота, 0,6%).
Общий процент представленности аминокислот белка Chn (рис. 5, 6) выявил, что в наименьшем количестве представлены аминокислоты His и Trp, самая большая представленность Ser, Leu, Val и Glu. В области каталитического центра также в меньшем числе встречаются His и Trp, но дополнительно снижается количество Asn. Каталитический центр обогащен аминокислотами – Gly, Leu, Ser, Phe, Thr. Особо необходимо отметить, что фенилаланин (Phe) в большей мере представлен именно в области каталитического центра, в сравнении с его встречаемости в общей протяженности белка. Следовательно, в общей аминокислотной последовательности Chn и в области каталитического центра в меньшей мере присутствуют положительно заряженные и ароматические аминокислоты, а в области каталитического центра снижается количество полярных незаряженных аминокислот. Также по всей протяженности белка отмечается большое количество полярных незаряженных, алифатических и отрицательно заряженных аминокислот. Тогда как в области каталитического центра, спектр аминокислот расширяется наряду, с алифатическими, полярными незаряженными, отмечаются нейтральные и ароматические (которыми обогащен каталитический центр).

Рисунок 2 - Выравнивание кодирующей нуклеотидной последовательности гена химозина исследуемых семейств позвоночных животных:
1 - R. norvegicus; 2 - T. chinensis; 3 - V. pacos; 4 - C. ferus; 5 - C. dromedarius; 6 - D. leucas; 7 - C. canadensis; 8 - A. alces; 9 - C. pygargus; 10 - B. bubalis; 11 - B. grunniens; 12 - B. taurus; 13 - O. aries; 14 - C. hircus
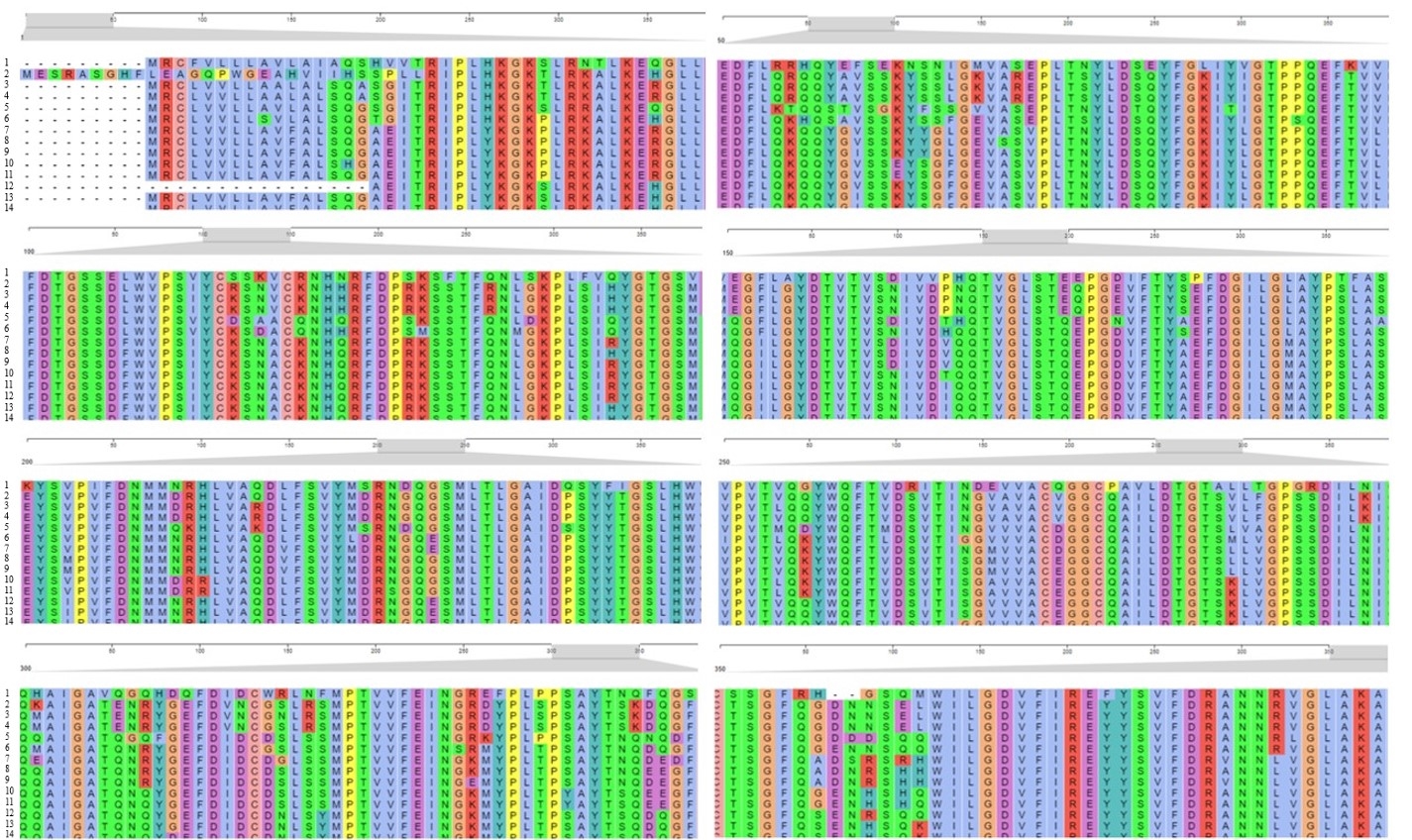
Рисунок 3 - Выравнивание аминокислотной последовательности гена химозина исследуемых семейств позвоночных животных:
1 - R. norvegicus; 2 - V. pacos; 3 - C. dromedarius; 4 - C. ferus; 5 - T. chinensis; 6 - D. leucas; 7 - C. canadensis; 8 - A. alces; 9 - C. pygargus; 10 - C. hircus; 11 - O. aries; 12 - B. bubalis; 13 - B. taurus; 14 - B. grunniens

Рисунок 4 - Консервативные участки (или отдельные аминокислоты) каталитического (активного центра) гена химозина исследуемых семейств позвоночных животных (выделены зеленым):
1 - C. dromedarius; 2- C. hircus; 3 - O. aries; 4 - B. taurus; 5 - R. norvegicus; 6 - C. ferus; 7 - B. grunniens; 8 - C. canadensis; 9 - D. leucas; 10 - T. chinensis; 11 - A. alces; 12 - C. pygargus; 13 - B. bubalis; 14 - V. pacos
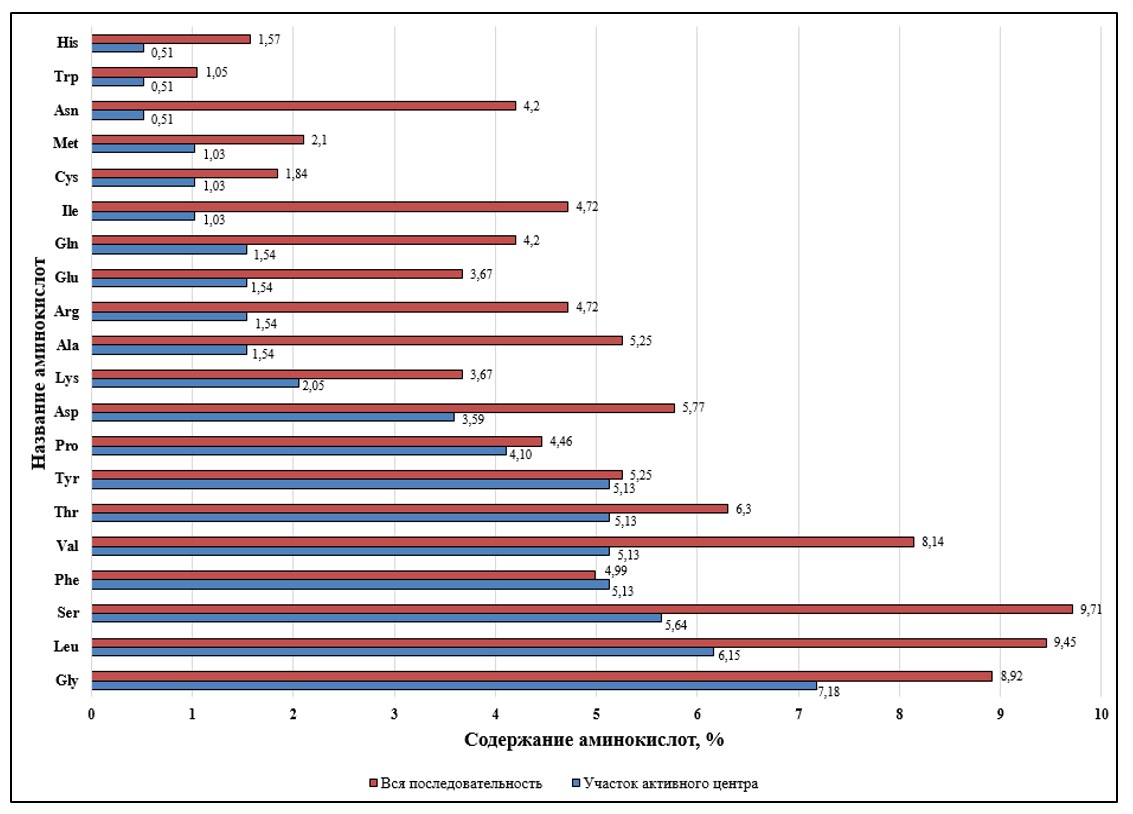
Рисунок 5 - Анализ содержания аминокислот в каталитическом центре и во всей аминокислотной последовательности химозина анализируемых семейств животных
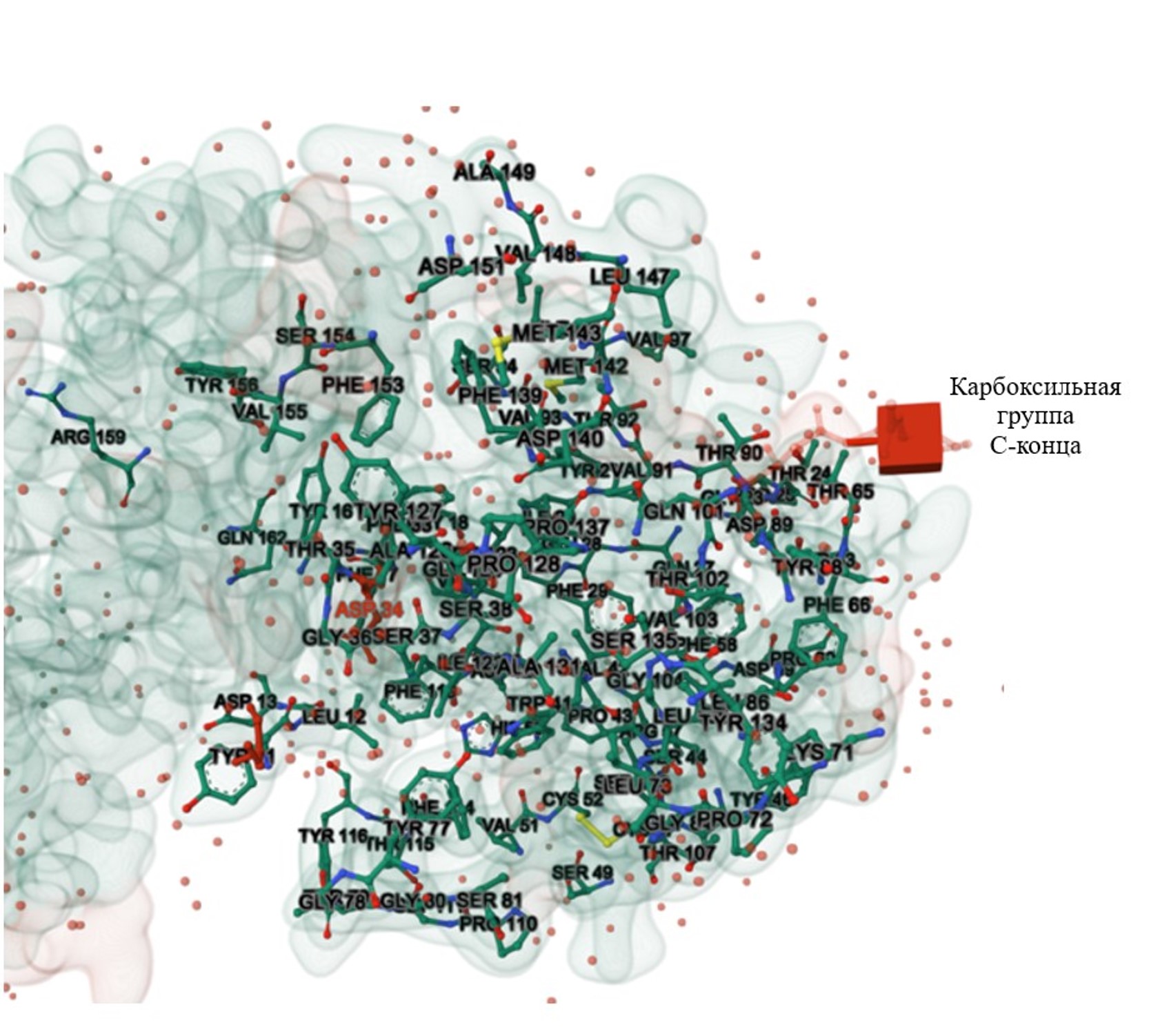
Рисунок 6 - 3D-модель каталитического центра белка химозина Camelus dromedaries с обозначением консервативныхучастков (синий и зеленые цвета)
- первая группа от 95-93% идентичности – отмечается между семействами Cervidae и Bovidae;
- вторая группа в диапазоне идентичности от 88-90% – отмечена у представителя семейства Delphinapterus leucas с Camelidae (C.ferus и C.dromedaries), Cervidae и Bovidae. А также у представителя семейства Camelidae с B.bubalis и B.Bosgrunniens (семейство Bovidae);
- третья группа в диапазоне идентичности от 85-87% – у представителей семейства Camelidae (C.ferus и C.dromedaries), с Cervidae и Bovidae. А также у V.pacos (семейство Camelidae) с B.Bosgrunniens (семейство Bovidae) и D.leucas;
- четвертая группа диапазон идентичности от 79-85% отмечен у T.chinensis (семейство Tupaiidae) с Camelidae, Cervidae, Bovidae и Delphinapterus leucas. А также V.pacos (семейство Camelidae) с Cervidae, Bovidae;
- пятая группа – идентичность представителя семейства Muridae (семейства R.norvegicus) в сравнении с другими семействами самая низкая – 74%.
Сравнительный попарный анализ аминокислотной идентичности (рис. 7) выявил различия с нуклеотидной. Так, в частности, нижний предел идентичности аминокислотной последовательности каталитического центра составляет 71-75%, верхний – 92-94%. Группы диапазона идентичности также отличаются:
- первая группа – 92-94%, полностью соответствует выявленной в первой группе нуклеотидной идентичности;
- вторая группа – диапазон идентичности составляет 85-87%. Также, как и в случае с нуклеотидной идентичностью, отмечено соответствие у представителя семейства Delphinapterus leucas с Camelidae, Cervidae и Bovidae. Отличием является то, что у представителей семейства Camelidae (C.ferus и C.dromedaries), аминокислотная идентичность выявлена не с B.bubalis и B.Bosgrunniens (семейство Bovidae), а с T.chinensis (семейство Tupaiidae) и Delphinapterus leucas;
- третья группа диапазон идентичности составляет 83-84%. Все представители семейства Camelidae (также, как и нуклеотидная идентичность) проявляют идентичность с Cervidae и Bovidae. А также отличительной особенностью является идентичность между T.chinensis (семейство Tupaiidae) и Delphinapterus leucas;
- четвертая группа диапазон идентичности от 81-82% отличием от выявленной нуклеотидной идентичности является лишь то, что у V.pacos (семейство Camelidae) идентичность выявлена с Delphinapterus leucas;
- пятая группа – идентичность 77-80% отмечена у V.pacos (семейство Camelidae) с Cervidae и Bovidae (кроме B.bubalis) и с T.chinensis (семейство Tupaiidae);
- шестая группа идентичность составляет 71-75% и полностью соответствует пятой группе по нуклеотидной идентичности гена Chn.
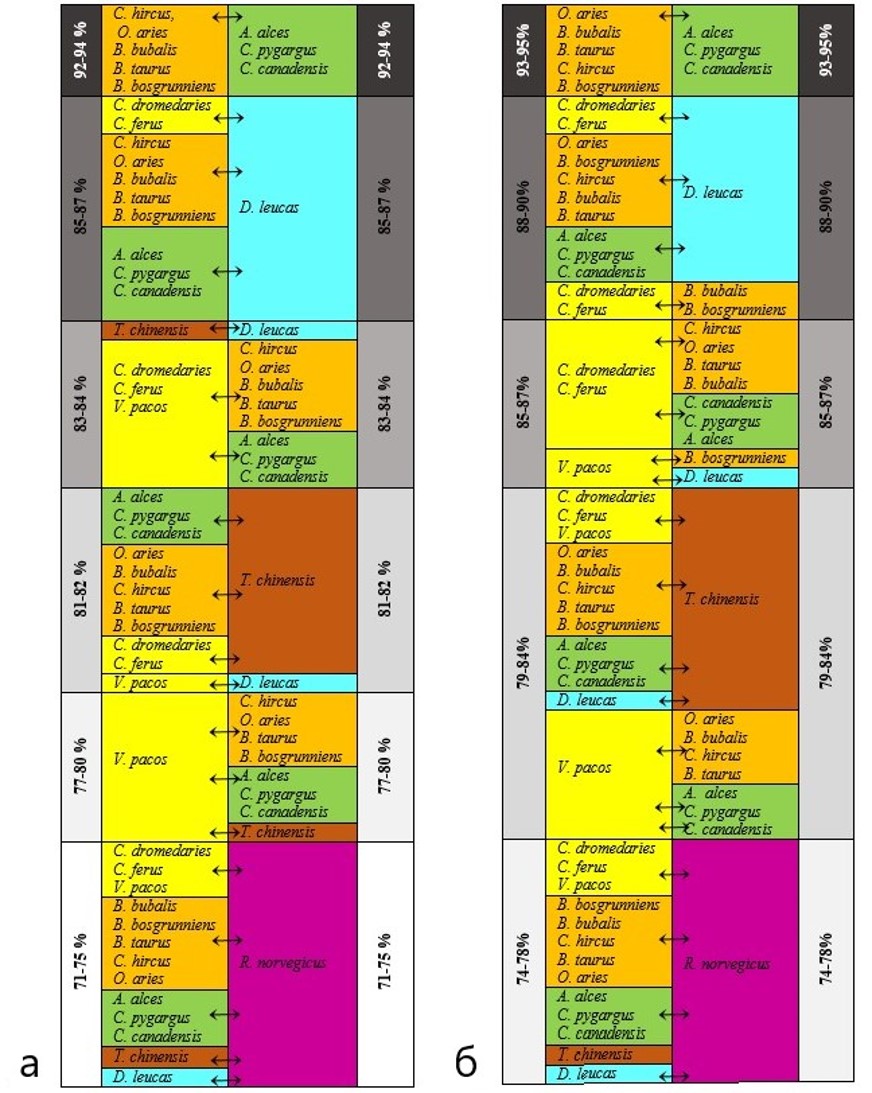
Рисунок 7 - Попарное сравнение аминокислотной (а) и нуклеотидной (б) идентичности каталитического участка химозина у представителей анализируемых семейств
Достаточно редко встречаются такие аминокислоты, как Trp (1,1-1,3%), Cys (1,5-1,9%), His (1,1-2,6%) и Met (1,8-2,9%). Самая редко встречающаяся ароматическая аминокислота – Trp, процентное содержание находится в пределах 1,1–1,3% и не проявляет выраженных отличий распределения у представителей анализируемых семейств. Серосодержащая аминокислота Cys также является редко встречающейся, содержание которой колеблется в пределах 1,8–1,9%, за исключением одного из представителей Camelidae (V. pacos – 1,5%) и Bovidae (B. bubalis – 1,6%). Положительно заряженные аминокислоты His в равной степени редко встречаются у представителей семейства Bovidae (1,1 – 1,6%), Camelidae (1,1 – 1,6%), Tupaiidae (1,3%) и большинства представителей Camelidae (1,6%, кроме V. pacos – 2,6%). У семейств Muridae и Delphinapterus leucas (R. norvegicus – 2,6% и D. leucas – 2,1%). Серосодержащая аминокислота Met отмечена у представителей семейства Cervidae (2,6 - 2,89%), Camelidae (V. pacos – 1,8%) и Muridae (R. norvegicus – 2,1%).
Относительно представленности других аминокислот среди представителей анализируемых семейств можно отметить, что положительно зараженная аминокислота Arg меньше всего отмечается у всех представителей Bovidae (2,4 – 3,2%), Tupaiidae (T. chinensis – 2,6%) и Delphinapterus leucas (D. leucas – 2,9%) и более всего у всех представителей Camelidae (4,4 – 4,7%) и Muridae (R. norvegicus – 4,5). Полярная незаряженная аминокислота Glu менее всего представлена у Tupaiidae (2,6%). Положительно зараженной аминокислоты Lys меньше всего у Tupaiidae (3,2%), Muridae (2,6%) и Delphinapterus leucas (2,9%). Такие аминокислоты алифатические как Asn, Pro, Ala, Ile и ароматическая Tyr не имеют ярко выраженных различий среди представителей анализируемых семейств. Тогда как ароматическая аминокислота Phe больше всего в каталитическом центре Chn у Muridae (7,1%), нейтральная аминокислота Gln – у Bovidae (6,3 – 7,1%) и Tupaiidae (7,1%), отрицательно заряженная аминокислота Asp и полярная незаряженная Thr – у Tupaiidae (7,4 – 7,6%).
Общий показатель консервативности по аминокислотному составу каталитического участка в положении 25 - 220 составляет 67% (118 идентичности из 175).
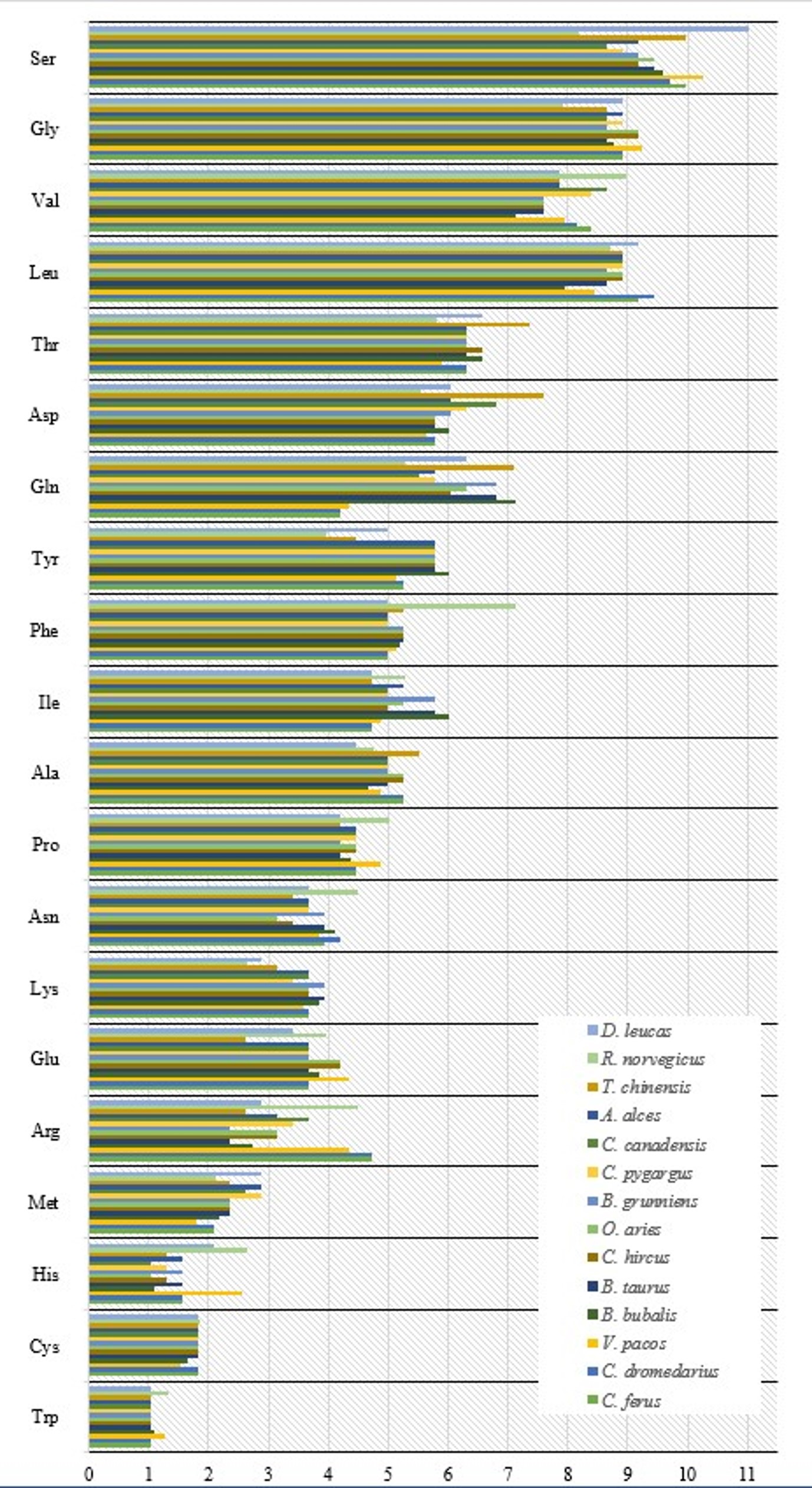
Рисунок 8 - Встречаемость аминокислот в области каталитического центра представителей анализируемых семейств
- группа I состоит из трёх видов семейства Camelidae (C. Dromedaries, Camelus ferus, Vicugna pacos);
- группа II включает три вида семейства Cervidae (A. Alce, C. Canadensis, C. pygargus);
- группа III – входят 5 видов семейства Bovidae, образующие две подгруппы.
В целом топология дерева трех групп соответствует топологии деревьев, построенных для других участков генома видов отряда Cetartiodactyla.
Отдельно стоят группы:
- группа IV – семейство Delphinapterus leucas;
- группа V – Muridae (семейства R.norvegicus)
- группа VI – T.chinensis (семейство Tupaiidae).
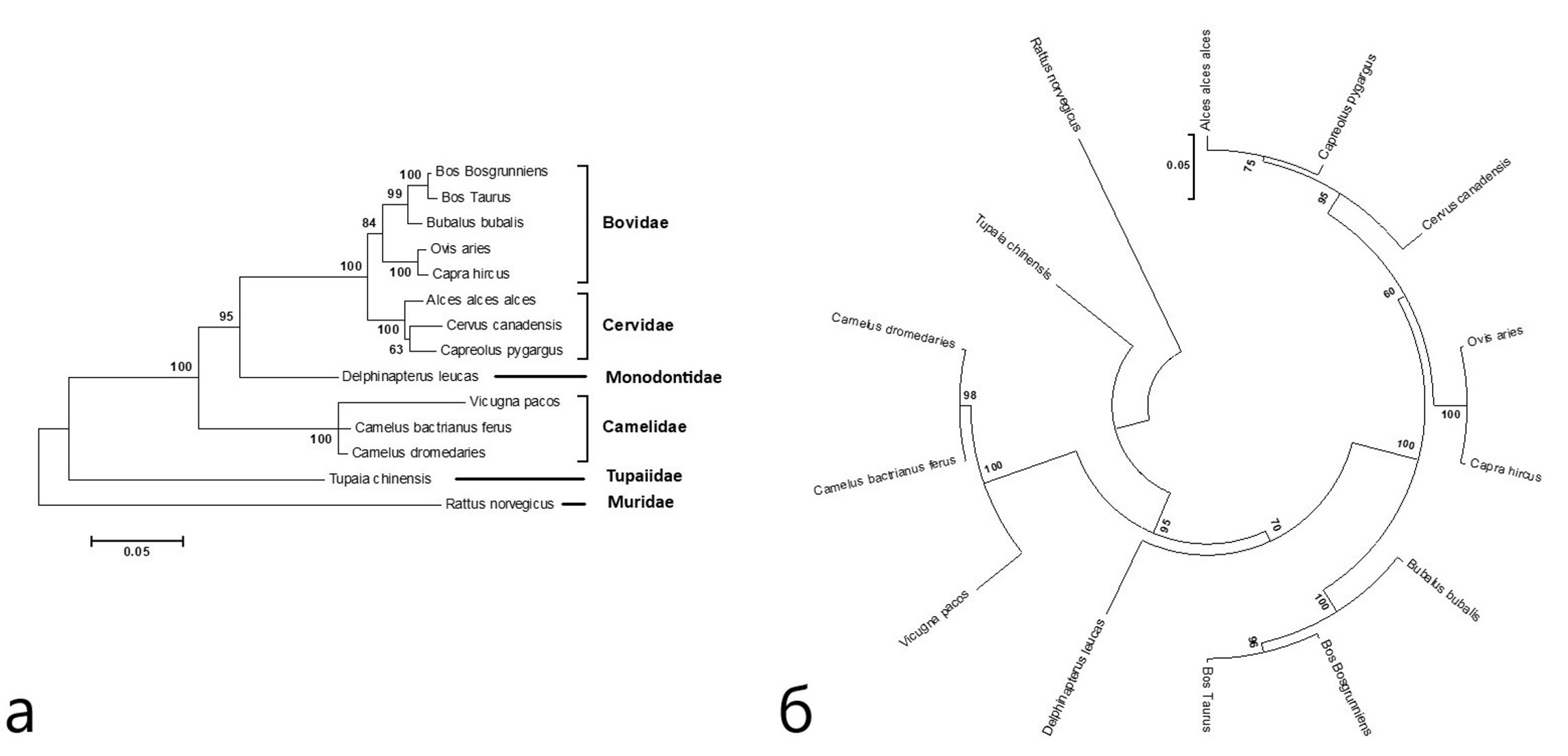
Рисунок 9 - Дендрограмма представителей анализируемых семейств построенная методом максимального правдоподобия на основе модели Tamura-Nei model (а) и метода присоединения соседей (Neighbor-Joining method) (б)
4. Заключение
Исследования Chn были сосредоточены главным образом на характеристике биохимических, структурных и функциональных свойств для промышленных целей , , , , в то же время анализ эволюционной динамики в ряду позвоночных аминокислотной и нуклеотидной последовательности крайне актуальна , , в связи с тем, что Chn был обнаружен у большого числа видов, как наземных, так и водных млекопитающих . Обоснованием для исследования белков заключается в механизмах мутаций, которые определяют изменения нуклеотидной последовательности гена и последовательно приводят к изменениям последовательности аминокислот в белке в процессе эволюции , , , . Так, в частности, дупликация и делеция генов, потеря генов и псевдогенизация , , , являются мощными драйверами эволюционных изменений, которые иллюстрируются изменчивым ферментативным спектром, участвующим в переваривании белков позвоночных включая Chn и являются реакцией на общие факторы окружающей среды, терморегуляции организма, рацион питания , соответственно рассматриваются в качестве ключевого фактора в формировании эволюционных изменений , , что определяется необходимостью в адаптивной фенотипической изменчивости с морфофункциональными последствиями для организмов , , . Более того, APS выполняют разнообразные функции от пищеварения до участия в формировании иммунитета , , , . Анализ генов Chn у разных млекопитающих выявил сходство в организации генов и эти результаты подтверждают мнение о том, что эти гены ферментов произошли от общего предкового гена .
Несмотря на то, что Chn демонстрирует высокую консервативность структуры и аминокислотных остатков в области каталитического центра , , , в ходе наших исследований выявлено, что наиболее консервативными остатками аминокислот и их позиции в области каталитического центра являются – Lys-Gly-K (26-28), Leu-Arg (30-31), Leu-Lys-Glu (34-36), Gly-Leu-Leu-Glu-Asp-Phe-Leu (38-44), Gln (47), Ser (52), Gly (59), Pro-Leu-Thr (65-67), Tyr-Leu-Asp (69-71), Tyr-Phe-Gly (74-76), Ile-Tyr (78-79), Gly-Thr-Pro (81-83), Gln-Glu-Phe (85-87), Val (89), Phe-Asp-Thr-Gly-Ser-Ser (91-96), Trp-Val-Pro-Ser (99-102), Tyr-Cys (104-105), Ser (107), Val-Cys (109-110), H (113), Arg-Phe-Asp-Pro (115-118), Ser (121), Thr-Phe (123-124), Lys-Pro-Leu (129-131), Tyr-Gly-Thr-Gly-Ser (135-139), Leu (142), Tyr-Asp-Thr-Val-Thr-Val-Ser (146-152), Ile-Val (154-155), Gln-Thr-Val-Gly-Leu-Ser-Thr (159-165), Pro-Gly (168-169), Phe-Thr-Tyr (172-173), Phe-Asp-Gly- Ile-Leu- ly (177-182), Ala-Tyr-Pro (184-186), Ala (188), Tyr-Ser (192-193), Pro (195), Phe-Asp (197-198), Met-Met (200-201), Leu-Val-Ala (205-207), Asp (209), Phe- Ser-Val-Tyr (211-214), Arg (217) и Gln (220). В белке в целом меньше всего встречаемость His и Trp, самая большая представленность Ser, Leu, Val и Glu. В области же каталитического центра, также, как и в общем в белке, в меньшей мере встречаются His и Trp, но дополнительно снижается количество Asn. Каталитический центр обогащен аминокислотами – Gly, Leu, Ser, Phe, Thr. Особо необходимо отметить, что фенилаланин (Phe) в большей мере представлен именно в области каталитического центра, в сравнении с его встречаемостью в общей протяженности белка.
Сравнительный анализ, после выравнивания, нуклеотидной и аминокислотной последовательности в области каталитического центра Chn у представителей анализируемых семейств выявил, что самая высокая идентичность отмечается в обоих случаях между семействами Cervidae, Bovidae и Camelidae (C. Dromedaries, Camelus ferus). Наименьшая, у семейства Muridae (семейства R.norvegicus) со всеми анализируемыми представителями семейств.
Выявлены специфические идентичности по нуклеотидной последовательности, которые формируются у Vicugna pacos (семейство Camelidae) с Bovidae, Delphinapterus leucas и Cervidae. Тогда как самая высокая идентичность по аминокислотной последовательности в области каталитического центра отмечена у Camelidae (C. Dromedaries, Camelus ferus) с T.chinensis (семейство Tupaiidae), Delphinapterus leucas. В свою очередь у семейства T.chinensis (семейство Tupaiidae) идентичны нуклеотидные последовательности с Delphinapterus leucas. У Vicugna pacos (семейство Camelidae) с Delphinapterus leucas. Наименьшая идентичность нуклеотидной последовательности отмечается у Vicugna pacos (семейство Camelidae) с Cervidae, Bovidae и T.chinensis (семейство Tupaiidae). Нижний предел идентичности нуклеотидной последовательности в области каталитического центра для анализируемых представителей семейств составляет 74%, а аминокислотной – 71%. Верхний предел идентичности нуклеотидной последовательности в области каталитического центра для анализируемых представителей семейств составляет 95%, а аминокислотной – 94%.
Представленность аминокислотного состава Chn носит выраженно видоспецифичный характер - достаточно редко встречаются Trp (1,1-1,3%), Cys (1,5-1,9%), His (1,1-2,6%) и Met (1,8-2,9%), часто – Ser (8,2-11,0%), Gly (7,9-9,2%), Val (7,1-9,0%) и Leu (8,0-9,5%). Общий показатель консервативности по аминокислотному составу каталитического участка в положении 25 - 220 составляет 67% (118 идентичности из 175).
В ходе филогенетического анализа на дендрограмме, построенной для нуклеотидной последовательности гена химозина, можно выделить клады шести клад:
- клада I состоит из трёх видов семейства Camelidae (C. Dromedaries, Camelus ferus, Vicugna pacos);
- клада II включает три вида семейства Cervidae (A. Alce, C. Canadensis, C. pygargus);
- клада III – входят 5 видов семейства Bovidae, образующие две подгруппы.
В целом топология дерева трех групп соответствует топологии деревьев, построенных для других участков генома видов отряда Cetartiodactyla.
Отдельно стоят группы:
- клада IV – семейство Delphinapterus leucas;
- клада V – Muridae (семейства R.norvegicus)
- клада VI – T.chinensis (семейство Tupaiidae).
Таким образом, выявлен широкий спектр как различий, так и сходства анализируемых параметров гена и белка Chn у исследуемых представителей семейств, которое по всей видимости определяется средой обитания, рационом питания, терморегуляцией организма и участием Chn в пассивном переносе материнского IgG, согласно «иммунной гипотезе». Выявленные замены, спектр и представленность аминокислот как в целом в белке, так и в области каталитического центра, могут влиять на связывание фермента с κCS и, следовательно, на каталитическую активность.
